 (153)
(153)
(昔プログラマの) プログラミング奮戦記(その2)
── JPEGファイルの解剖と解析
▼JPGファイルの解読
先の「昔プログラマの プログラミング奮戦記」で紹介したようにデジタルカメラに記録された画像データから、付随する情報を読み取るプログラムをPerl言語で記述しようと試みてきた。この種のファイルはJPEGファイルと呼ばれているが、いろいろな形式がある。ここでは、Exifファイルフォーマットと呼ばれ規格化されているものを対象としている。
▼Perlプログラムでの処理
先に紹介したプログラム(*1)の中で使われている GetJPGsize という関数を使うことにより、画像の横の長さと縦の長さを求めることができるようになった。
これが使えると、手作業によらないで沢山の画像ファイルに対し一括して適用できるようになり大変な省力化になる。しかし残念ながらこの関数の記述では、複数のJPGファイルに対し繰り返し適用しようとすると2度目以降はうまく動作しないという不都合が発生してしまう。つまりこの関数では、繰り返し同じ機能を発揮してくれないのである。
プログラミングの世界では、こういう状況を「reusableでない」と言う。reusableでない関数は、全くとは言わないまでもほとんど利用価値がない。 【注】(*1)これまで、GetJPGsize.pl というファイル名にしてきたが、関数名とファイル名を区別するため、以後ファイル名は AnalizeJPG.pl とする。
不都合が発生する原因をいろいろと追究してきたが、原因は未だに解明されていない。文字コードを扱うのが専門のPerlという言語で、無理やり処理しているのが原因なのかもしれない。Perlでこの種のデータをただやみくもに読み込んだりすると、データの一部が壊される可能性があるからである。たとえばデータの中に改行コードがあると、Perl処理系は実行環境に合わせて変換するのが普通である。
Perl では改行は '\n' と表現されるので,一文字から成るように見えるが、実行環境によっては1文字(CR)の場合、あるいは2文字(CR+LF)の場合と違いがある。それぞれに実行環境に合うよう自動的に置き換えられる。したがってただ無暗に読み込んだりすると、たまたま制御コードと同じパターンのデータがあれば、誤って変換され結果としてデータが壊されてしまうことになる。そういうことにならない様、ここではライブラリ関数の read ではなく sysread の方を用いることにした。更に、データ構造をしっかりと確認し、画像を構成する本来のデータ部分はできるだけ触れない(読まない)ようにすることにした。
▼JPGファイルの構造を学び直す
ここで、JPGファイルの構造をしっかりと学び直すことにした。
インターネット上で「JPGファイルの構造」とか「Exifファイルフォーマット」を指定して検索すれば有益な情報が得られる。
現時点で公表されている資料では、Exifファイルフォーマットのバージョンは v2.1 と記されているものが最新のようである。残念ながら私の使っているカメラでは、Exifファイルフォーマットはv1.1であるから古い仕様であることが分かった。
素人カメラマンの私にとって、ファイルフォーマットが古いからと言ってそう簡単にカメラを買い替える訳にはいかない。したがってここでの説明は最新の仕様に沿うものではないので、もしかすると互換性がない部分があるかもしれない。以下は考え方だけを理解してもらえばよいので簡単な説明で済ますことにしよう。
▼解剖と解析
方針として、先ず全体の「解剖」(JPGvivisect.pmで処理)を行って、その後で個々に「解析」(AnatomyJPG.plで処理)するという手順を取ることにした。ただし、呼び出しの手順は AnatomyJPG.pl の処理の前段階で JPGvivisect.pm を呼び出すようになっている。

(図1:処理の手順)
JPGファイルでは、本来の画像データの前に情報セグメントが複数個置かれている。
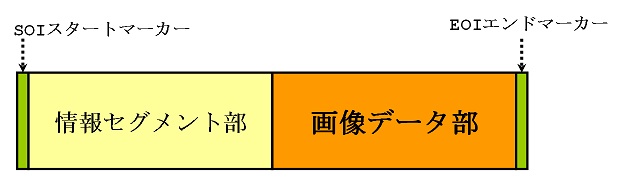
(図2:JPGファイルの構造)
この「情報セグメント部」に属する「各情報セグメント」の「種類」と「位置関係」を明確にする必要がある。つまり解剖して各セグメント(内臓)の種類と位置と寸法が分かるようにする。そして全体の骨格が明確になった後で、初めて個々のセグメントの中を解析することにする。
▼解剖プログラム(JPGvivisect.pm)
解剖プログラムは最も基本的なプログラムなのでパッケージ化し、ライブラリとして使えるようにしてある。
この「解剖結果」を記憶する場所として %HashTBL という名のグローバルなハッシュ変数を用意する。グローバル変数なので他のモジュールからもアクセスできるようになっている。
各セグメントは先頭にはマーカー(2バイト)があり、次に長さ情報(2バイト)が置かれている。この値がマーカーに続く情報部分(セグメントパラメータ部)の長さである。つまりこの2バイトの長さを置く場所も情報部分に含まれる。

(図3:セグメントの構造)
$HashTBLというハッシュ変数をどのように使うかを説明しよう。
ハッシュ変数というのは、<キー>と<値>を対にして記憶するものであるが、$HashTBLでは<キー>として JPGファイルのセグメント情報を示す「マーカー」を利用する。<値>としては、そのマーカーの<位置情報>を記憶する。
(表:代表的なマーカーの一覧)
| Merker | Code |
| SOI(Start of Image) | FFD8 |
| APP0(JFIF) | FFE0 |
| APP1(Exif) | FFE1 |
| APP1(Exif) | FFE1 |
| DQT(Define Quantization Table) | FFDB |
| DQT(Define Quantization Table) | FFDB |
| SOF0(Start Of Frame 0) | FFC0 |
| DHT(Define Huffman Table) | FFC4 |
| DHT(Define Huffman Table) | FFC4 |
| DHT(Define Huffman Table) | FFC4 |
| DHT(Define Huffman Table) | FFC4 |
| SOS(Start Of Scan) | FFDA |
| EOI(End Of Image) | FFD9 |
ただし、同じマーカーが繰り返し登録される可能性もあるので、<位置情報>には登録個数とそれぞれの位置情報を保存できるようにする。そのため、値はリスト形式にしてある。
( [0], [1], [2], [3], .... )
[0]に登録個数、[1]以降に位置情報が置かれる。
最初に
$HashTBL{ <キー> }[0];
とするとマーカーが%HashTBLに登録され、値を置く位置の[0]はまだ空であるがPerlでは空はゼロと解釈されるので、
$i = ++$HashTBL{ <キー> }[0];
と書いておけば [0]の値が 1 に、$i にも 1 が代入される。
次の行の
$HashTBL{ <キー> }[$i] = <位置情報>;
で<キー>に対応する<位置情報>が[$i]つまり[1]の位置に保存される。
以下同様にして、登録のたびに[0]に個数が
それぞれの位置情報が[1],[2],[3],[4],...
に保存されていく。
▼解析プログラム(AnatomyJPG.pl)
解析段階では、この解剖段階で得られた情報を使って一般の利用者(カメラ愛好家)が必要とするであろう各種情報を得られるようにする。得られた「解析結果」は %DataTBL という名の、これもグローバルなハッシュ変数上に保存し共用されるようにする。
解析結果を記憶したければ、適当に識別名を付けて値を代入すればよい。
$DataTBL{'File Name'} = "$fileString";
例えば、SOF0('FFC0')マーカーに属するデータを得たければ
$pos =$HashTBL{'FFC0'}[1];
として'FFC0'マーカーの位置情報を取り出し getByte関数を使ってセグメントパラメータ部から値を取り出すことができる(第2引数が1なら文字型データを、2以上ならそのバイト数の数値データとして取り出される)。
$h = getByte($pos+5,2);
$w = getByte($pos+7,2);
これらを $DataTBLに保存するときは、数値にコンマを挿入(insCom関数)したり、数値の単位も合わせて記録しておくと利用するときに便利である。
$DataTBL{'横幅'}=insCom($w)." Pixel\n";
$DataTBL{'高さ'}=insCom($h)." Pixel\n";
詳しい技法については、
解析プログラム(AnatomyJPG.pl)
や
解剖プログラム(JPGvivisect.pm)
を参照してください。


|